
Simen Rød Sandve er en av flere forskere som har kartlagt hvetens DNA. Det kan bety bedre og mer robuste sorter i fremtiden.
(Foto: Håkon Sparre, NMBU)
Hvete har det største og mest kompliserte genomet av alle kornsortene.
Arbeidet med å kartlegge det, har vært betydelig mer vanskelig enn det var for eksempel for ris og mais.
Seks ganger større enn menneskets
– For å sette det i perspektiv, er hvetens genom seks ganger større enn menneskets, sier Odd-Arne Olsen ved NMBU – Norges miljø- og biovitenskapelige universitet.
Årsaken til brødhvetens store genomstørrelse ligger i hvordan arten oppstod.
- Les også: Forsker frem framtidas hvete
Brødhvete oppstod igjennom to naturlige hybridiseringer, det vil si at ulike arter krysset seg med hverandre. Det betyr at brødhveten faktisk består av DNA fra tre ulike arter. Den har derfor tredoble sett med kromosomer. 21 istedenfor 7.
– Dette har gjort arbeidet med kartleggingen av DNAet ekstra komplisert. Vi har måttet skille DNA fra tre ulike sett med arvemateriale, forteller Olsen.
– Det er først når vi kjenner hvetens DNA – når vi har sekvensert hvetegenomet – at vi har mulighet for å foredle nye hvetesorter på en mer effektiv måte.
Et 70 år langt arbeid
Hveten er blant verdens mest produserte planter.
Foredling av hveten har blitt gjort av forskere langt tilbake i tid. På 1950-tallet jobbet blant annet forskere ved Norges landbrukshøgskole med å forbedre kvaliteten og produksjonen .
Norsk hvete hadde lenge for dårlig kvalitet til å kunne bli brukt til bakemel, men dette ble sakte, men sikkert, endret gjennom systematisk arbeid av norske hveteforedlere.
- Les også: Bedre bakst med hvetegenom
Nye sorter ble utviklet og dyrkingsteknikkene forbedret. I de beste årene på 90-tallet kunne så mye som 80 prosent av norsk hvete brukes til baking.
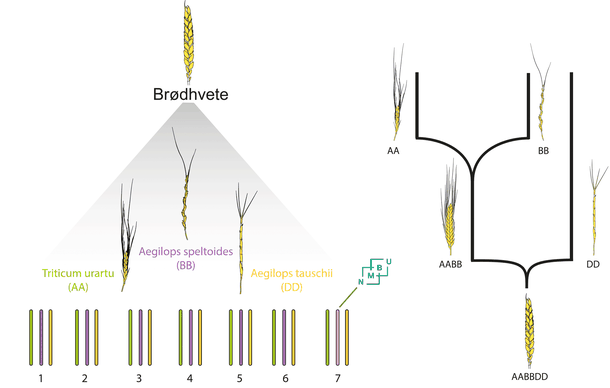
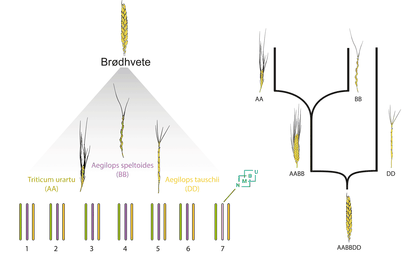
Brødhvetens genom. Til venstre: Brødhvetens kromosomer fra tre ulike nært beslektede arter. Triticum urartu, Aegilops speltoides, Aegilops tauschii er de nærmest beslektede artene til brødhvetens tre kromosomsett. Til høyre: De to naturlige hybridiseringene som er opprinnelsen til brødhvete.
(Illustrasjon: NMBU)
Dramatisk dårligere hvete
Både resistens for groskade og egenskaper for glutenproteiner har blitt forbedret gjennom forskning gjennom flere år.
Dessverre har de siste årene med meget spesielle klimaforhold ført til dramatisk nedgang i norsk hvetekvalitet.
– Målet med hveteforskningen er å sikre en mer effektiv tradisjonell foredling av nye hvetesorter som tåler mer ekstreme værforhold, som mer tørke og mer nedbør, sier Olsen.
– Det er også et mål å få hvetesorter med bedre bakekvalitet og høy ernæringskvalitet.

Professor Odd-Arne Olsen har ledet arbeidet med å sekvensere brødhvete ved NMBU.
(Foto: Håkon Sparre, NMBU)
I disse dager er de norske forskerne medforfattere i hele tre av fem artikler i forskningsjournalen Science, hvorav to publiseres fra NMBU med utenlandske medforfattere.
Alle fem artiklene handler om kartleggingen av hvete-DNAet. Forskere fra Asia, Nord-Amerika og Europa har bidratt i arbeidet, gjennom IWGSC (International Wheat Genome Sequencing Consortium).
- Les også: Hvete sliter med væte og tørke
Ansvar for kromosom 7B
Hvetegenomet består altså av 21 kromosomer. Forskerne ved NMBU har hatt ansvaret for å sekvensere ett av disse: kromosom 7B. Olsen har ledet prosjektet, og forsker Simen Rød Sandve er en av fem forskere fra NMBU i prosjektet.
Å sekvensere kromosom 7B har vært et nitid arbeid som har pågått over år.
– Ironisk nok har DNA-sekvensen til kromosom 7B og de resterende 20 hvetekromosomene liten verdi i seg selv, sier Rød Sandve.
– Det er ikke før man får identifisert gener og forstår hvordan disse genene gir seg til uttrykk i plantens egenskaper, at genomsekvenser virkelig blir verdifulle.
Mer robuste sorter
– Resultatene vi nå har kommet frem til, kan føre til at vi kan utvikle sorter som er spesialtilpasset ulike klima. Veien videre er å aktivt utnytte den genetiske informasjonen i foredlingsprosessen, sier Rød Sandve.
Et av de viktigste bruksområdene vil være å identifisere genetiske markører for sykdomsresistens og deretter overføre resistensgener mellom foredlingsmateriale og nye hvetesorter.
Referanser:
Matthias Pfeifer m.fl.: Genome interplay in the grain transcriptome of hexaploid bread wheat. Science. Juli 2014. DOI: 10.1126/science.1250091
Thomas Marcussen m.fl.: Ancient hybridizations among the ancestral genomes of bread wheat. Science. Juli 2014. DOI: 10.1126/science.1250092
Kellye Eversole m.fl.: Slicing the wheat genome. Science. Juli 2014. DOI: 10.1126/science.1257983

Leave a Reply